Biologie Santé.
Interaction avec la biologie et la médecine
Thématiques :
- Mécanobiologie - Contact : Rachele Allena, rachele.allena@univ-cotedazur.fr
- DREAMS NEMATIC (réseaux biologiques) - Contact : Yves D'Angelo, Yves.Dangelo@unice.fr
- Modélisation et Imagerie de la mobilité séminale en vue de la conception de tests de fertilité automatisés - Contact : Didier Auroux, auroux@unice.fr
- Réseaux de régulation intracellulaires - Contact : Elisabeth Pécou-Gambaudo, elisabeth.pecou@univ-cotedazur.fr
- Imagerie médicale, neurosciences (spikes) - Contact : Elena Di Bernardino, elenadb@unice.fr
- Simulation numérique d'un système complet d'imagerie médicale micro-onde - Contact : Victorita Dolean, victorita.dolean@unice.fr
- Problèmes de transmission d'information dans les cellules - Contact : Thierry Goudon, goudon@unice.fr
- Modèles de populations structurées pour la croissance tumorale et pour la polymérisation (maladies à prions, Alzheimer, etc.) - Contact : Thierry Goudon, goudon@unice.fr
- Modélisation de la dynamique des adipocytes - Contact : Thierry Goudon, goudon@unice.fr
- Modélisation du système respiratoire - Contact : Benjamin Mauroy, mauroy@unice.fr
- Simulation numérique de modèles liés aux accidents vasculaires cérébraux (AVC) - Contact : Stéphane Descombes, sdescombes@unice.fr
- Croissance bactérienne - Contact : Jean-Baptiste Caillaux, jean-baptiste.caillau@univ-cotedazur.fr
- Classification de données appliquée à la détection de maladies - Contact : Jean-Baptiste Caillaux, jean-baptiste.caillau@univ-cotedazur.fr
- Simulation numérique de la migration uni et pluricellulaire et rôle du noyau - Contact : Rachele Allena, rachele.allena@univ-cotedazur.fr
- Simulation numérique du remodelage osseux - Contact : Rachele Allena, rachele.allena@univ-cotedazur.fr
- Institut NeuroMod (Neurosciences) - Contact : Patricia Reynaud-Bouret (reynaudb@unice.fr)
- Contact : Rachele Allena, rachele.allena@univ-cotedazur.fr
- Membres : Rachele Allena, Benjamin Mauroy, Olivier Pantz
- Partenaires : InPhyNi, IRCAN, IBV, LP2M, INRIA, IPMC
- Résumé : La mécanobiologie étudie la réponse des cellules et des tissus à leur environnement mécanique au cours de différents processus biologiques tels que la cancérogenèse ou l’embryogénèse. Au cours des 30 dernières années, des avancées majeures ont permis d’observer in vitro le comportement des cellules et plus particulièrement comment elles se déforment sous une contrainte spécifique (i.e. traction ou pression), comment elles réorganisent leur cytosquelette ou déclenchent des réponses moléculaires et/ou chimiques. D’un côté ces nouvelles techniques expérimentales et d’imagerie ont permis d’avancer, de l’autre côté les observations sont encore souvent qualitatives car il est très difficile de quantifier les contraintes, les déformations ainsi que les paramètres mécaniques associés à un comportement cellulaire spécifique. La modélisation mécanique et mathématique joue alors un rôle fondamental car elle permet d’explorer un grand nombre de scénarios avec un temps et un coût moindres comparés aux essais en laboratoire. Ce projet implique pour l’instant des C et EC du LJAD, de l’InPhyNi, du LPMC, de l’IRCAN, ainsi que des chercheurs hors UCA.
DREAMS NEMATIC (réseaux biologiques)
- Membres : R. CATELLIER, Y. D'ANGELO, T. GOUDON, C. GUERRIER, L. MONASSE
- Partenaires : LIED PARIS (Florence CHAPELAND-LECLERC, Pascal DAVID, Éric HERBERT, Gwenaël RUPRICH-ROBERT ) MAP5 PARIS (Amandine VEBER)
CMAP (Milica TOMASEVIC) - Résumé : Le projet DREAMS porte initialement sur la modélisation multi-échelles de réseaux biologiques tels que les champignons filamenteux. Dans le cadre de l'ANR NEMATIC, le projet implique des C et EC de 4 équipes du Laboratoire ainsi que des probabilistes, physiciens, biologistes de 2 autres laboratoires parisiens, LIED et MAP5. In vivo, le champignon modèle Podospora anserina évolue dans un environnement hautement compétitif, où plusieurs dizaines d’espèces sont présentes et se nourrissent de matériel végétal partiellement dégradé. La croissance de P. anserina dans ce milieu confiné semble être fortement contrainte par l’accès à des ressources limitées ou parcellaires, com- binés à une concurrence féroce avec d’autres organismes. Comprendre comment ces champignons ont pu concevoir des réseaux de transport efficaces pour s’étendre et se densifier reste très mal connu. Notre objectif est d’explorer les caractéristiques- clés du processus d’expansion de P. anserina sous diverses contraintes et à différentes échelles, du niveau cellulaire au niveau macroscopique, à la fois par des moyens numériques et des réalisations expérimentales à l’échelle du laboratoire.
Réseaux de régulation intracellulaires
- Membres : E. PÉCOU-GAMBAUDO, S. GIREL, Y. D'ANGELO, T. GOUDON
- Résumé : Les modèles mathématiques de la dynamique de ces réseaux de régulation basés sur les lois de la cinétique des réactions biochimiques traduisent uniquement les flux d’information à travers la cellule. Ceci est une vue partielle sur l’organisation intracellulaire car on sait que l’environnement physique intracellulaire y joue un rôle important. Comprendre comment la physique contribue au maintien de la vie de la cellule est la motivation initiale de notre projet de recherche. On cherche à mettre en évidence les mécanismes de régulation intracellulaire faisant intervenir des processus physiques, et à en produire des modèles mathématiques simples. Notre point de départ consiste à représenter la physique dans les réseaux de régulation à travers l’enthalpie libre des réactions, autrement dit, il s’agit pour nous d’étudier la thermodynamique des réseaux de régulation. Un point qui nous intéresse particulièrement concerne les transitions de phases conduisant à la création d’organelles sans membranes ayant diverses propriétés régulatrices (e.g. le blocage de la traduction des ARN retenus dans ces organelles).
Modélisation et Imagerie de la mobilité séminale en vue de la conception de tests de fertilité automatisés
- Membres : Gilles Aubert, Didier Auroux
- Partenaires : Pierre Degond (IMT, Toulouse), Laure Blanc-Féraud (I3S), Xavier Druart
(INRA Tours), Franck Plouraboué (IMFT, Toulouse) - Mots-clefs : modélisation, apprentissage, motilité massale, flot optique
- Résumé : L’objectif de ce projet est de développer les concepts nécessaires à la réalisation de tests automatisés de fertilité d’échantillon de semences animales. L’observation au microscope des échantillons de semence met en évidence une turbulence induite par le mouvement d’ensemble des spermatozoïdes, dite motilité massale. Il a été établi que la motilité massale est un excellent critère de fertilité de l’échantillon (alors que la motilité individuelle ne l’est pas). Le projet a pour but de développer des techniques d’imagerie et de scoring qui permettront d’automatiser la sélection des échantillons fertiles et le rejet des autres par l’observation de cette motilité. Ces techniques seront dans un deuxième temps améliorées par l’utilisation de modèles adéquats. La modélisation des mécanismes collectifs en jeu dans le mouvement d’ensemble de la semence pourra donner des pistes pour comprendre pourquoi la motilité individuelle des spermatozoïdes n’est pas bien corrélée avec la motilité massale.
Simulation numérique d'un système complet d'imagerie médicale micro-onde
- Contact : Victorita Dolean (victorita.dolean@unice.fr)
- Membres : V. Dolean, F. Rapetti, R. Pasquetti
- Partenaires : C. Pichot, J.-Y. Dauvignac, C. Migliaccio, I. Aliferis (LEAT, UNSA); F. Nataf, F. Hecht (LJLL, Paris 6), M. de Buhan (MAP5, Paris 5)
- Mots-clefs : équations de Maxwell, problèmes inverses, imagerie médicale micro-onde, simulation numérique (méthodes numériques d'ordre eleve, décomposition de domaine, calcul parallèle).
- Résumé : L'imagerie micro-onde par tomographie est une nouvelle modalité d'imagerie permettant une détection dans une phase précoce de développement des tissus malades, avec un certain nombre d'applications médicales potentiellement attractifs. Avec la tomographie par micro-ondes dans la gamme de fréquences de plusieurs centaines de MHz à quelques GHz, les tissus sont bien différenciées et, par conséquent peuvent être imagés, sur la base de différences dans les propriétés diélectriques (c.-à-contraste). Il a été prouvé dans de nombreuses études que les propriétés diélectriques des tissus biologiques sont un bon indicateur de leurs conditions fonctionnelles et pathologiques. Du point de vue mathématique cela suppose la modelisation de l'interactions onde-matiere en champs proche, la diffusion des ondes électromagnétiques arbitraires dans des milieux très hétérogènes, sur une large plage de fréquences dans le domaine des micro-ondes et le développement méthodologique et numérique d'un nouvel outil d'inversion robuste, associé à la solution numérique du problème électromagnétique directe. Les applications envisagées ici seront les suivantes: imagerie micro-onde par tomographie d'un AVC (accident vasculaire cérébral), des lésions cérébrales, imagerie fonctionnelle cardiaque, … à partir de données synthétiques et expérimentales en collaboration avec une société d'imagerie médicale électromagnétique.
Problèmes de transmission d'information dans les cellules
- Contact : Thierry Goudon (goudon@unice.fr)
- Membres : Gilles Aubert, Didier Auroux
- Partenaires : R. Blossey, Jean-François Bodart (Institut de Recherche Interdisciplinaire, Lille), P. Lafitte (ECP)
- Mots-clefs : réaction-diffusion, MAPK, exposant de Hill.
- Résumé : L’enjeu de ce travail consiste à prendre en compte les aspects spatiaux dans des modèles de propagation de signaux à travers le cytoplasme. Plus précisément on s'intéresse à un système de type réaction-diffusion généralisant la dynamique de Michaelis-Menten pour la cascade MAPK dans les oeufs de Xenope. Cette étude met en évidence certaines conditions portant sur les données initiales et au bord pour assurer le phénomène de propagation à l’intérieur de la cellule. On discute aussi le rôle de paramètres définissant la dynamique chimique, comme l'exposant de Hill.
Modèles de populations structurées pour la croissance tumorale et pour la polymérisation (maladies à prions, Alzheimer, etc.)
- Contact : Thierry Goudon (goudon@unice.fr)
- Membres : Gilles Aubert, Didier Auroux
- Partenaires : M. Doumic (Inria Rocquencourt), P. Lafitte (ECP)
- Mots-clefs : EDP, populations structurées, coagulation and break-up
- Résumé : On s'intéresse à divers systèmes d'EDP décrivant l’évolution de populations structurées. Ces modèles, plus ou moins directement inspirés de la science des matériaux, peuvent décrire des phénomènes biologiques comme la croissance de certaines tumeurs ou les mécanismes à l’oeuvre dans des maladies neuro-dégénératives, comme les maladies à prions. L'enjeu consiste à étudier le comportement qualitatif des solutions, notamment pour caractériser les états asymptotiques en temps longs, directement accessibles à l’expérience, et à analyser leur stabilité. Un autre volet est consacré à la conception de méthodes numériques spécifiques pour ces problèmes.
Modélisation du système respiratoire
Modélisation appliquée aux pathologies pulmonaires et à la kinésithérapie respiratoire
- Contact : Benjamin Mauroy (mauroy@unice.fr)

- Membres : Didier Clamond, Riccardo Di Dio, André Galligo, Thomas Laporte, Benjamin Mauroy, Valentin Mons, Bernard Rousselet, Jonathan Stéphano
- Partenaires : P. Flaud (MSC, Université Paris 7), J. Merckx (médecin anesthésiste, spécialiste des enfants, ex-Hôpital Necker), M. Argentina (INLN) ; Dominique Pelca et Christian Fausser (Kinésithérapeutes libéraux, Paris) ; Angelos Mantzaflaris (INRIA Sophia); Cyril Karamaoun (LJLL, Paris) ; Barrett Mitchell, Michaël Brunengo (société RespInnovation Sophia) ; L. Giovanini-Chami, M. (Hôpital Lenval) ; C. Delclaux, P. Bokov (Hôpital Robert Debré, Paris) ; C. Barthod, L. Goujon, M. Loubet (SYMME, Chambéry) ; A. Decoesne (IMB, Bordeaux) ; S. Martin (MAP5, Paris)
- Mots-clefs : kinésithérapie respiratoire, systèmes d’EDO ou d’EDP de grande dimension, éléments finis
- Résumé : La kinésithérapie respiratoire joue un rôle très important dans le traitement de maladies pulmonaires comme la bronchiolite chez les bébés ou encore la mucoviscidose. Elle consiste à imposer des pressions sur le thorax pour expulser les mucosités en excès qui tapissent les bronches. Cependant, les manipulations effectuées par les kinésithérapeutes sont empiriques et différentes pratiques existent, certaines mettant en avant l'efficacité du geste (haut débits pendant un temps court) qui rend la manipulation plus incommodante pour le patient alors que d'autres mettent en avant le confort du patient (bas débits pendant un temps long) mais ont une efficacité controversée. La modélisation de l'expectoration assistée peut donc donner des indications permettant de guider le praticien dans son geste et aider à améliorer les appareils d’expectoration mécanique. La modélisation du comportement des bronches et des mucosités mène à des systèmes d’EDO ou d’EDP de grandes dimensions, qui sont couplées aux équations de la mécanique dans le thorax.
Etude de la géométrie des organes assurant le transport de l'oxygène, évolution
- Contact : Benjamin Mauroy (mauroy@unice.fr)
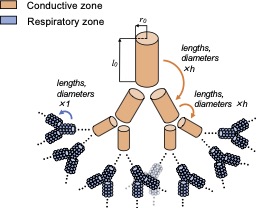
- Partenaires : Y. Privat (LJLL, Paris 6); P. Dantan, P. Flaud (MSC, Paris 7); B. Mahut, C. Delclaux, P. Bokov (HEGP, Paris); Frédérique Noël (INRIA Paris)
- Mots-clefs : équations de Navier-Stokes, équations de convection-diffusion-réaction, interaction fluide-structure, optimisation
- Résumé : Nous cherchons à mieux comprendre les échanges et le transport de l'oxygène chez les mammifères et les optimisations sous-jacentes du poumon et du réseau vasculaire induites par l'évolution (lois allométriques, médecine darwinienne). Nous développons des modèles mathématiques et numériques multi-échelles depuis l'organe (bronches par exemple) jusqu'à la cellule (typiquement le globule rouge). Nous étudions les échanges d'oxygène à la barrière air/sang et les réactions chimiques entre l'oxygène et l'hémoglobine dans le globule rouge.
Modélisation de la morphogenèse du système respiratoire : bronches et réseau vasculaire
- Contact : Benjamin Mauroy (mauroy@unice.fr)
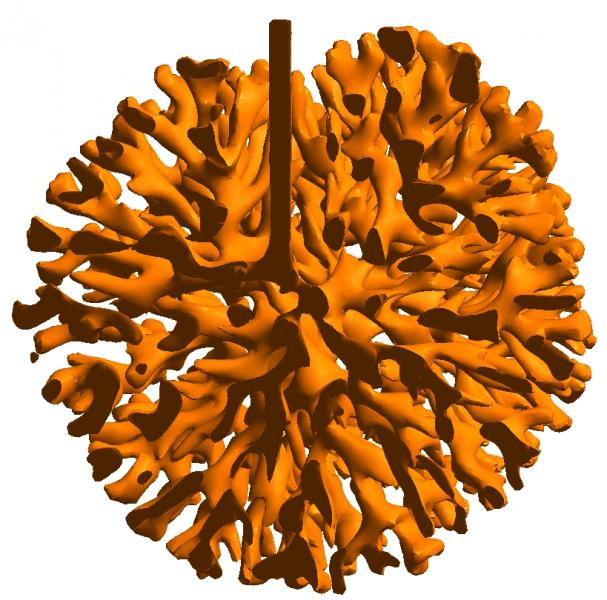
- Membres : Benjamin Mauroy, Marc Monticelli
- Partenaires : Stéphane Douady et Annemiek Cornelissen (Biophysique, MSC Paris 7); Pierre Blanc et Vincent Sapin (CHU Clermont-Ferrand); Raphaël Clément (IBDM, Marseile)
- Mots-clefs : morphogenèse, instabilité, poumon, réseau sanguin, éléments finis
- Résumé : Le poumon des mammifères se développe pendant l'embryogenèse à partir d'une bronche initiale entourée par le mésenchyme pulmonaire. La paroi des bronches croît dans le mésenchyme pulmonaire et subit des branchements répétés jusqu'à former l'arbre adulte. Parallèlement dans le mésenchyme, se forment les artères bronchiques, suivant précisément les bronches (direction, branchements, etc). Nous développons un modèle mathématique et numérique du développement des bronches et des artères bronchiques. Ce modèle est basé sur une instabilité qui émerge quand les bronches croissent avec le flux d'un facteur croissance induit par un laplacien.
Sites web :
Simulation numérique de modèles liés aux accidents vasculaires cérébraux (AVC)
- Contact : Stéphane Descombes (sdescombes@unice.fr)
- Partenaires : équipe NACHOS (INRIA Sophia-Antipolis)
- Résumé : Le domaine de la biologie ciblé est la médecine et les outils mathématiques utilisés sont les méthodes numériques performantes et rapides pour les équations de réaction-convection-diffusion.
Etude des caractéristiques géométriques d’ensembles aléatoires d’excursion pour l’imagerie médicale
- Contact : Elena Di Bernardino (elenadb@unice.fr)
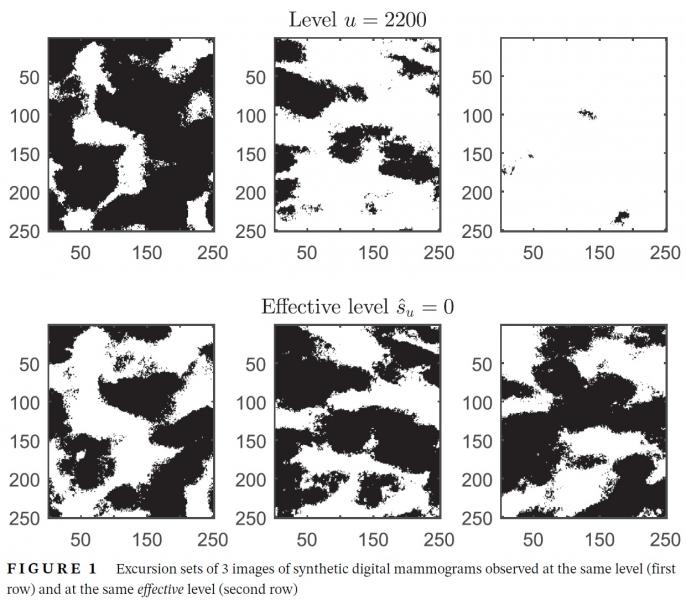
- Partenaires : M. Abaach (Ph.D, LJAD - Université de Paris), H. Biermé (Université de Tours), C. Duval (Laboratoire Painlevé, Université de Lille), A. Estrade (MAP5 Université de Paris)
- Mots-clefs : Géométrie aléatoire, ensembles d’excursion, imagerie médicale, mesures de courbure de Lipschitz-Killing
- Résumé : Nous cherchons à mieux comprendre les aspects géométriques des images en travaillant avec les mesures de courbure de Lipschitz-Killing de leur ensembles d’excursion. Cela nous permet de construire des tests ainsi que de comparer des images entre elle à l’aide des méthodes adaptatives. De plus, ces caractéristiques géométriques nous permettent de segmenter nos images enfin d’en extraire des éléments spécifiques (masses, opacités, frontières entre tissus etc). Différentes applications sont en cours sur des mammographies (synthétiques et réelles) ainsi que sur des radiographies pulmonaires.
- Membres : Jean-Baptiste Caillau, Jean-Baptiste Pomet, Agustin Yabo
- Partenaires : Walid Djema (Inria, Sophia), Hans Geiselmann (U. Grenoble), Jean-Luc Gouzé (Inria, Sophia), Hidde de Jong (Inria, Grenoble), Sofya Maslovskaya (U. Paderborn)
- Mots-clés : self-replicator models, maximisation du taux de croissance, bioingénierie
- Résumé : la croissance de micro-organismes est essentiellement un problème d’optimisation qui consiste à allouer dynamiquement des ressources à telle ou telle fonction de la cellule, de façon à maximiser un taux de croissance ou un autre critère biologique. Les modèles dynamiques, dits « self-replicators », sont utilisés pour formuler ce problème dans le cadre du contrôle optimal et permettent de rendre compte d’observations in vivo. Les questions de contrôle qui en résultent sont souvent délicates, à cause des non-linéarités des modèles, de la présence de paramètres incertains, de la coexistence de différentes échelles de temps, d’un environnement en constante évolution, et de la présence de nombreuses contraintes physiques ou chimiques. Le projet Maximic (financement ANR) cherche à répondre à ces questions en étudiant les stratégies d’allocation de ressources des micro-organismes, et en proposant de nouvelles lois de contrôle basées sur des procédés de bioingénierie (notamment optogénétique).
- Membres : Jean-Baptiste Caillau
- Partenaires : Michel Barlaud (I3S, Sophia), Antonin Chambolle (Dauphine), Cyprien Gilet (I3S)
- Mots-clefs : classification supervisée et non supervisée, sélection de paramètres, optimisation parcimonieuse, single cell data
- Résumé : la disponibilité de données génétiques de patients (atteints de cancers, notamment), en particulier grâce au séquençage single cell, permet de recueillir des informations dont la structure est bien spécifique : les cohortes sont de quelques centaines d’individus, chacun étant représenté dans un espace de très grande dimension, de l’ordre de 10^4 gènes. Les techniques de réduction de dimension sont donc critiques dans ce contexte, si on souhaite pouvoir automatiser la classification entre individus sains et malades. Un objectif tout aussi important, à des fins de médecine préventive, est d’arriver à sélectionner les gènes discriminants dans cette classification. L’optimisation parcimonieuse joue un rôle majeur de ce point de vue.
Simulation numérique de la migration uni et pluricellulaire et rôle du noyau
- Contact : Rachele Allena
- Mots clés : EDP , réaction-diffusion, éléments finis, mécanique du noyau
- Résumé : Nous développons des modèles numériques pour simuler la migration d’une ou plusieurs cellules dans différentes configurations telles que la durotaxis ou le confinement en 2D et 3D. Nous nous intéressons en particulier au rôle du noyau, l’organelle la plus volumineuse et la plus rigide de la cellule, qui peut avoir un rôle déterminant lors de l’invasion cellulaire à travers des pores de taille sub-nucléaire.
Simulation numérique du remodelage osseux
- Contact : Rachele Allena
- Mots clés : EDP , éléments finis, mécanotransduction, micro-endommagement
- Résumé : Le remodelage osseux est un phénomène mécano-biologique qui assure un renouvellement constant d’un os sain et est initié par la mécano-transduction des ostéocytes. Il peut être perturbé par une pathologie qui génère du micro-endommagement responsable de l’apoptose ostéocytaire et donc d’un déséquilibre entre résorption et apposition d’os. Nous développons des modèles numériques capables de coupler l’environnement mécanique à l’activité des différents phénotypes cellulaires impliqués et de prendre en compte l’influence du micro-endommagement sur la viabilité ostéocytaire.
- Analogue hydrodynamique de la phyllotaxie - Contact : Christian Mathis, mathis@unice.fr
- Modélisation de la prolifération des algues Ostreopsis en Méditerranée - Contact : Magali Ribot, ribot@unice.fr
- Modélisation du mouvement des fibroblastes sur un scaffold - Contact : Magali Ribot, ribot@unice.fr
- Migration de cellules endothéliales sur des substrats attractifs - Contact : Magali Ribot, ribot@unice.fr
- Modélisation de la croissance de biofilms de cyanobactéries se développant au fond de fontaines - Contact : Magali Ribot, ribot@unice.fr
